Mapas de ativação e testes para fMRI
Para esse exercício, vamos utilizar dados de uma tarefa do tipo stroop. Essa tarefa consiste na medida do tempo de reação e na acurácia da leitura, em voz alta, de palavras em duas situações: 1. As palavras estão escritas na mesma cor que a cor expressa pelo significado semântico - denominamos por CONGRUENTES 2. As palavras estão numa cor que difere da cor expressa pelo significado semântico (exemplo: a palavra vermelho impressa com tinta azul) - denominamos por INCONGRUENTES
Estima-se que na segunda situação ocorre um atraso no processamento da cor da palavra, induzido pelo conflito no processamento da informação e por conseguinte, causando tempos de reação mais lentos e um aumento de erros.
Quando se trata de experimentos, o maior erro dos novatos é acreditar que com um estímulo só é possível comparar repouso com o estímulo. É necessário ter uma amostra representativa para conseguir garantir os dados para comparação.
Para análise, seguiremos os seguintes passos:
- Definição do block design do experimento
- Coleta de dados
- Pré-processamento do sinal: Correção do motion correction (quando a pessoa se mexe durante a aquisição de dados. Nesse caso é feito o realinhamento, com corpo rígido: 3 parâmetros rotação, 3 parâmetros de translação e/ou combinações desses movimentos).
- Especificar a convolução da HRF - hemodynamic response function (veremos adiante)
- Aplicação do GLM no sinal BOLD para cada voxel x.y.z, usando como variável preditora os dados de convoluídos
- Aplicação de um teste estatístico para definir quais voxels são relevantes para construção dos mapas
- Visualização e armazenamento dos mapas
A ideia aqui é investigar a ativação cerebral para cada uma das situações: CONGRUENTES e INCONGRUENTES. Então, primeiramente realizamos a leitura dos dados:
# Imagem esta preprocessada
# SPM (UCL) e FSL (Oxford)
require(AnalyzeFMRI)## Carregando pacotes exigidos: AnalyzeFMRI## Carregando pacotes exigidos: R.matlab## R.matlab v3.6.2 (2018-09-26) successfully loaded. See ?R.matlab for help.##
## Attaching package: 'R.matlab'## The following objects are masked from 'package:base':
##
## getOption, isOpen## Carregando pacotes exigidos: fastICA## Carregando pacotes exigidos: tcltk## Warning in fun(libname, pkgname): couldn't connect to display ":0"##
## Attaching package: 'AnalyzeFMRI'## The following object is masked from 'package:RNifti':
##
## orientation#Leitura de dados de fMRI
volume=f.read.volume("./data/Stroop.nii")
#Leitura das condicoes (desenho experimental)
congruente=scan("./data/congruent.txt")
incongruente=scan("./data/incongruent.txt")Obs: O comando scan é melhor (mais rápido) para ler vetor do que o read.table, mais usado para ler .txt
A função resposta ao estimulo congruente (ativação durante o estimulo, CONG = 1; e não ativação, fora do estímulo CONG = 0), como se observa em:
ts.plot(congruente)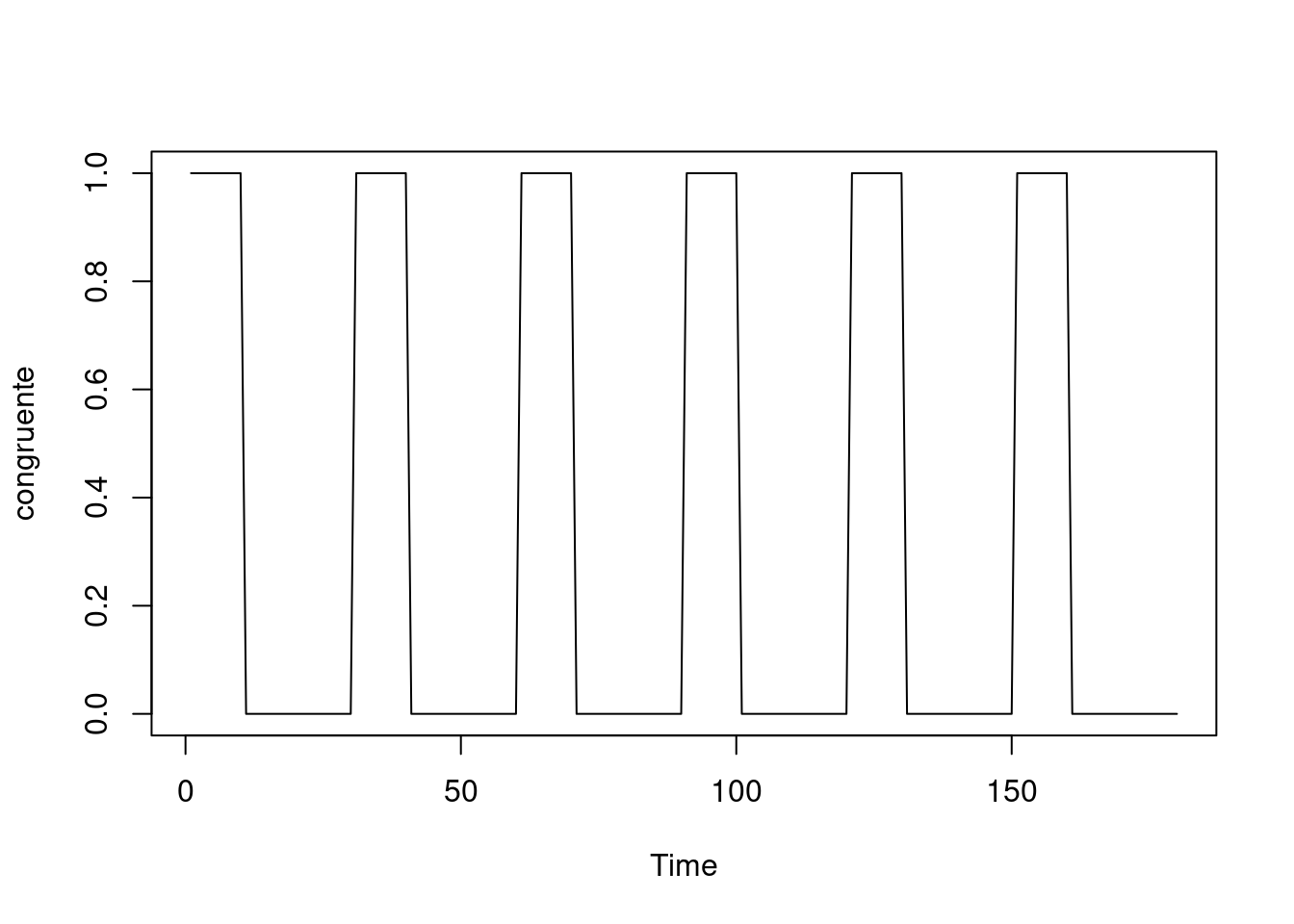
O objetivo da análise é estimar se, e em que medida, cada preditor contribui para a variabilidade observada no curso do tempo do voxel. Considere, por exemplo, uma experiência na qual a resposta BOLD,
Realizemos agora a convolução dos estímulos sobre a resposta hemodinâmica esperada do indivíduo. Ou seja, ao ser submetido ao estimulo, espera-se que ocorra uma ativação cerebral associada a essa resposta. Essa ativação chamada de função resposta hemodinâmica (HRF – hemodinamic response functional).
A equação abaixo mostra a convolução da função resposta pela HRF pela HRF do modelo de Garry Glover:
Em código, essa função dada pelo modelo de Garry Glover faz duas curvas gaussianas e a HRF é a combinação linear dessas duas gaussianas. Os parâmetros já seguem por default:
glover=function(HZ){
a1=6
a2=12
b1=0.9
b2=0.9
d1=5.4
d2=10.8
c=0.35
x=seq(0, 30, 1/HZ) # HZ is the Sampling Rate (Heartz)
glover1=((x/d1)^a1)*exp((-x+d1)/b1)
glover2=((x/d2)^a2)*exp((-x+d2)/b2)
G=glover1-c*glover2
return(G)
}Determinando HRF: Função de resposta hemodinâmica, considerando um tempo de repetição TR de 2s, ou seja 1/2:
#HRF- Funcao de resposta hemodinamica
HRF = glover(0.5)Exemplo de event-related (similar ao potencial evocado, vários experimentos coletados coincidindo). No caso de estímulos muito consecutivos, a curva basal nem retorna ao original, o que dificulta na visualização:
CONDICAO = array(0, 90)
CONDICAO[30] = 1
CONDICAO[60] = 1
ts.plot(CONDICAO)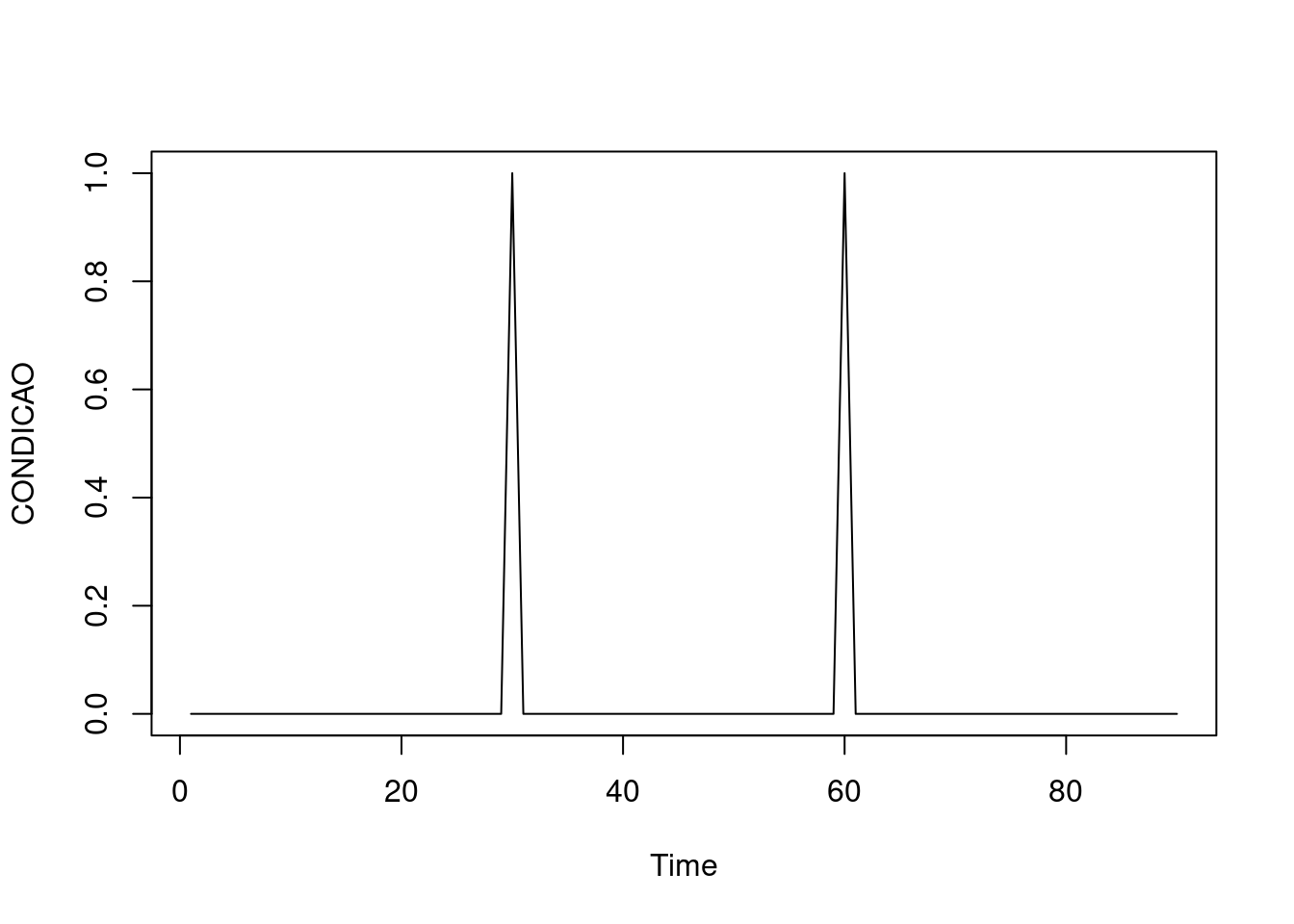
# Convolucao do vetor condicao pela HRF
# ATENCAO: NAO LER o pacote signal, pois a função
# que faz a convolução tem o mesmo nome da que faz a
# filtragem em frequência
X = filter(CONDICAO, HRF, sides=1, method="convolution")
ts.plot(cbind(CONDICAO, X), col = c(1,2))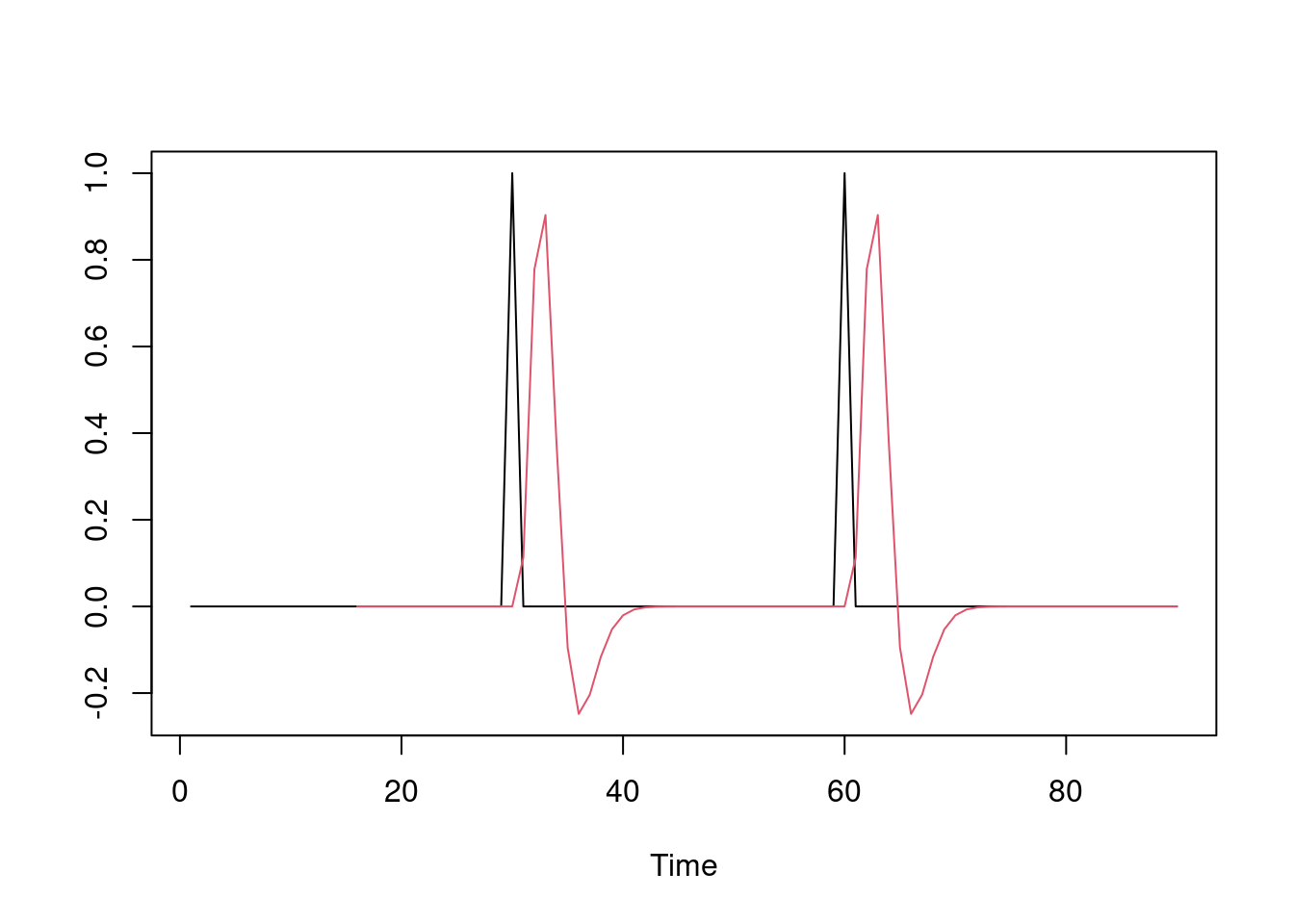
Exemplo de Desenho block-design:
CONDICAO = array(0, 90)
CONDICAO[20:40] = 1
CONDICAO[60:80] = 1
X = filter(CONDICAO, HRF, sides=1, method="convolution")
ts.plot(cbind(CONDICAO, X), col=c(1, 2))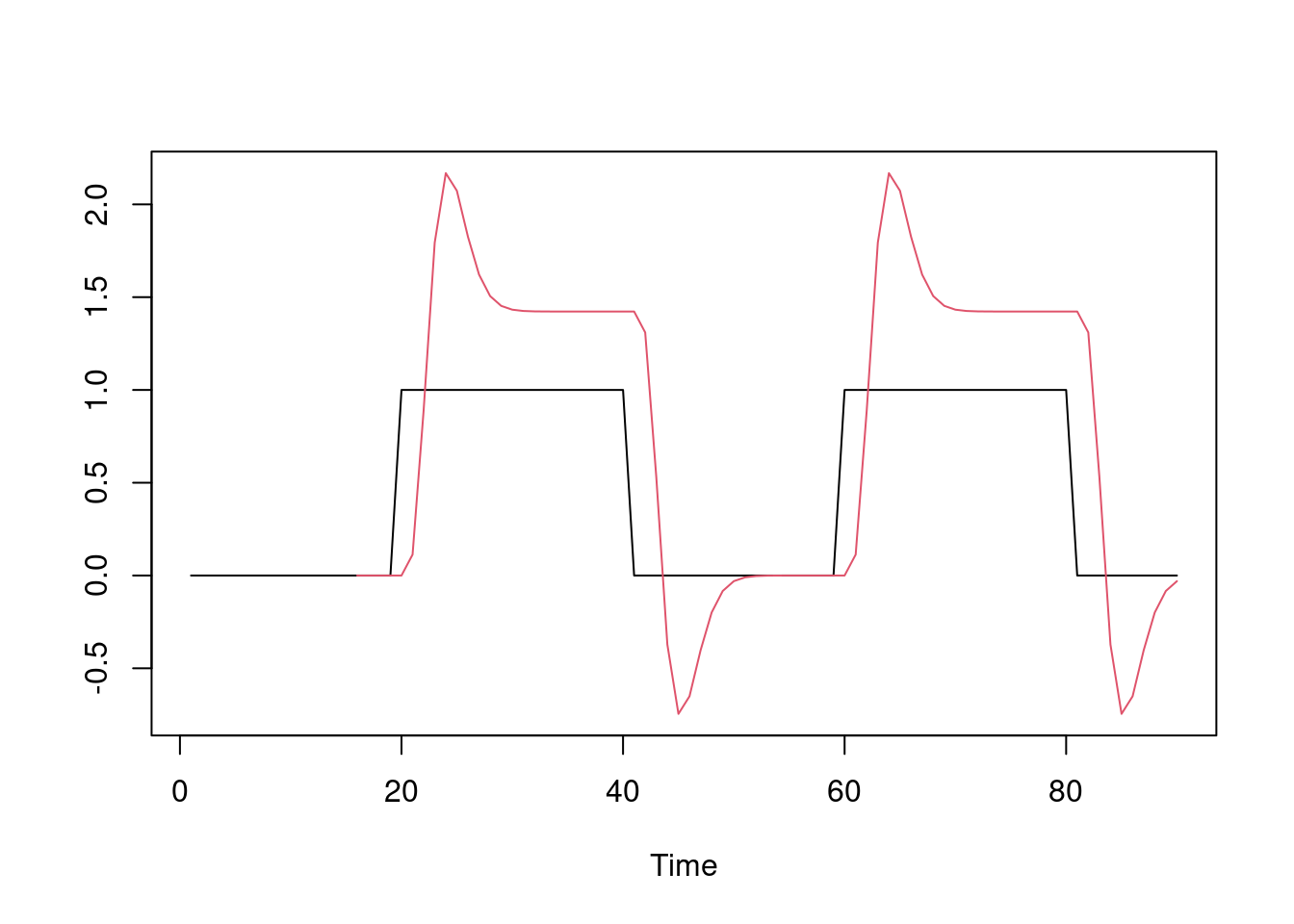
Identificando a convolução das condições pela HRF:
#Convolucao da condicao congruente pela HRF
X=filter(congruente, HRF, sides=1, method="convolution")
#Convolucao da condicao incongruente pela HRF
Z=filter(incongruente, HRF, sides=1, method="convolution")Ao plotarmos a função que representa um modelo de ativação relacionado ao design do experimento, verificamos comportamentos semelhantes. Analisando as duas condições:
ts.plot(cbind(congruente, incongruente), col=c(1,2))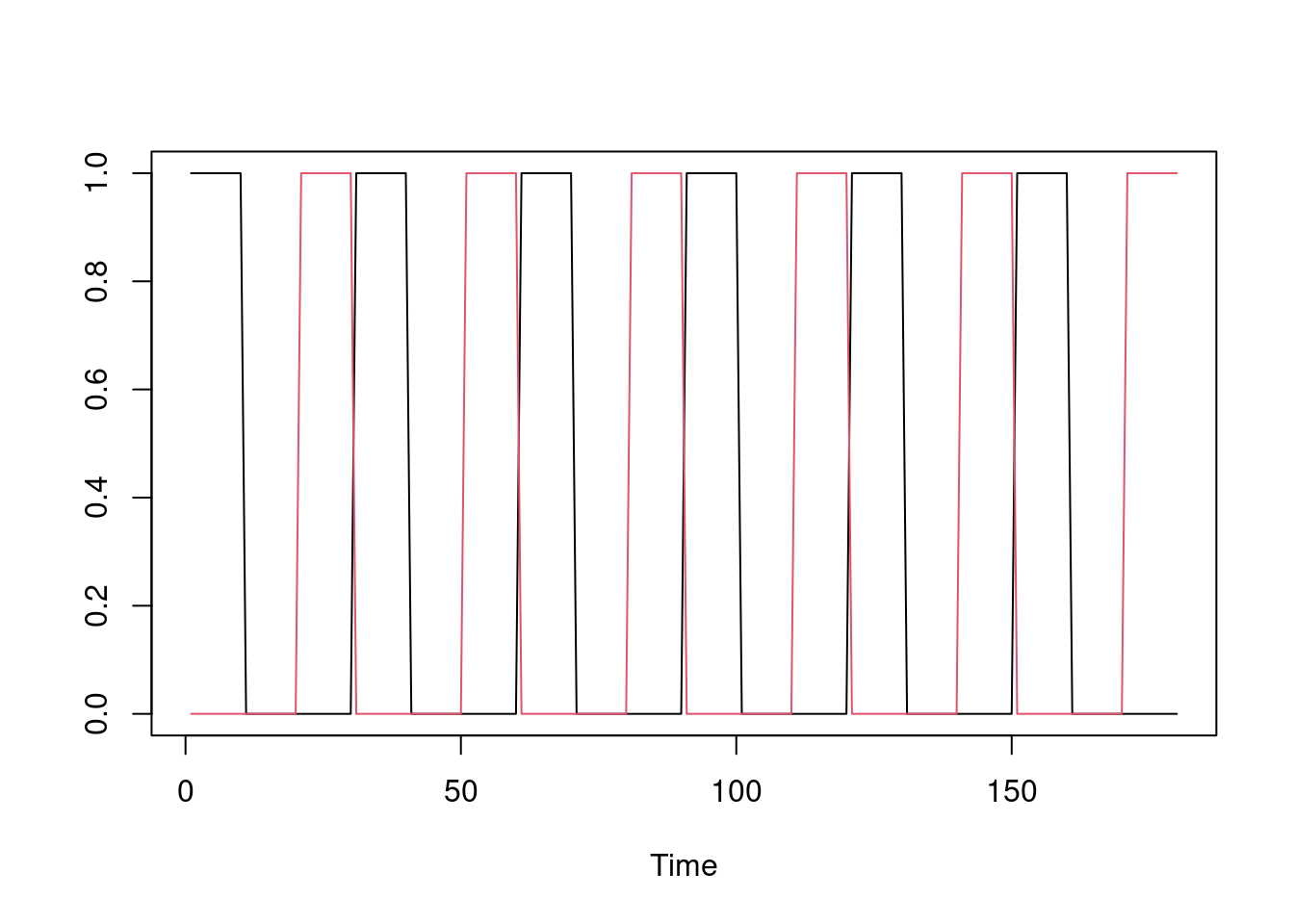
ts.plot(cbind(X, Z), col=c(1, 2))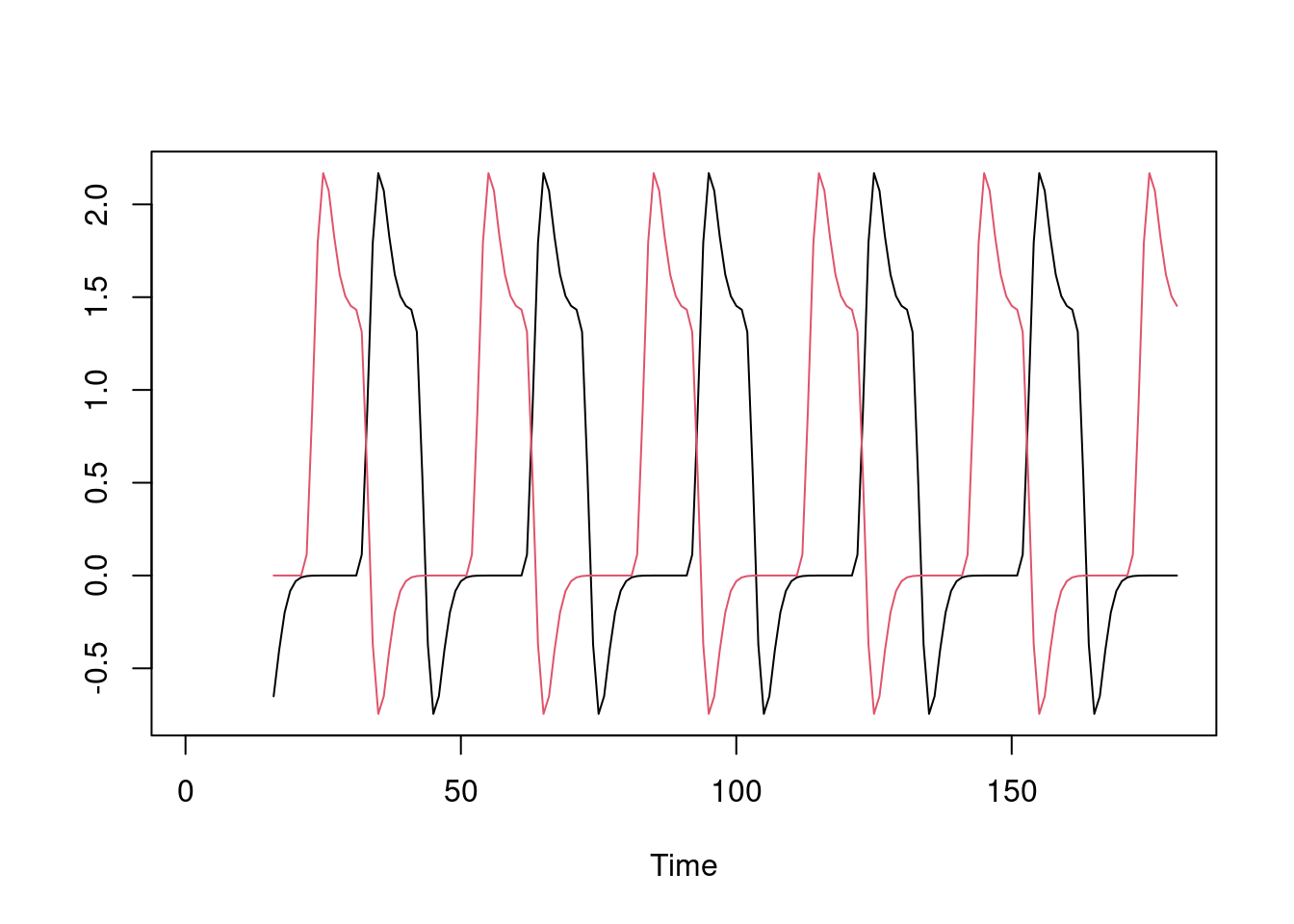
A título de curiosidade, essa convolução utilizada na função filter é equivalente ao seguinte código (as saídas são equivalentes):
#Faça a convolução do INCONG pela HRF.
Z = array(0,length(incongruente))
for(ti in (length(HRF)+1):length(incongruente)){
for( i in 1:length(HRF)){
Z[ti]= Z[ti]+ incongruente[ti-i]*HRF[i]
}
}
#Convolução do CONG pela HRF
X = array(0,length(congruente))
for(ti in (length(HRF)+1):length(congruente)){
for( i in 1:length(HRF)){
X[ti]= X[ti]+ congruente[ti-i]*HRF[i]
}
}
#Normalizando (colocar o máximo em 1) os dados da condição para facilitar a interpretação.
X = X/max(X)
Z=Z/max(Z)Para garantir uma resposta hemodinâmica mais próxima do que se espera é feita uma convolução da resposta esperada relacionada ao desenho do experimento com a função resposta hemodinâmica do modelo de Garry Glover, dando origem a um sinal convoluído que representa como seria, idealmente, o comportamento do sinal BOLD para aquele modelo de experimento.
Realizando o ajuste do modelo linear geral:
No GLM, faremos uma regressão linear múltipla onde o Y é o sinal BOLD, usando a HRF convoluída. No fundo, quero saber se o sinal se comporta conforme a ativação do voxel (se o voxel é ativado por um determinado estímulo). Para cada voxel x, y e z vamos rodar o GLM (Regressao multipla) usando como variável preditora os dados dessa convolução.
Para regressão múltipla, faremos um ajuste de nível (
neste caso, supondo:
Para comparação, faremos um teste de hipóteses. Analisando pelo t-valor, estatística t (verificar ativa ou não e se é estatisticamente diferente de zero ou não): Estatística T do beta correspondente a condição congruente é igual ao do modelo.
Obs: O valor p não armazena sinal, ou seja, não conseguimos verificar existência de ativação ou de desativação.
Fazendo os Mapas de estatisticas T:
# Verificando a dimensão do volume para identificar como serão os mapas:
dim(volume)## [1] 45 54 45 180Considerando no caso do GLM o
# Considera as dimensões da matriz volume e adiciona esse 1, para construção do mapa
mapaTcongruente = array(0,c(45,54,45,1))
mapaTincongruente = array(0,c(45,54,45,1))
for(xi in 1:45){
for(yi in 1:54){
for(zi in 1:45){
#pega somente os voxels intracranianos, ou seja, diferentes de zero:
if(volume[xi,yi,zi,1]!=0){
#Ajuste do modelo linear geral - GLM:
modelo=lm(volume[xi,yi,zi,]~X+Z)
#Estatistica T do beta correspondente a condicao congruente
mapaTcongruente[xi,yi,zi,1] = summary(modelo)$coef[2,3] # Beta_1
#Estatistica T do beta correspondente a condicao incongruente
mapaTincongruente[xi,yi,zi,1] = summary(modelo)$coef[3,3] #Beta_2
}#fecha if
}}}#fecha for do xi,yi,ziSe quero testar se
Se quero testar se
Se quero testar se
E por fim, se quero testar a ativação Incongruente > Congruente, ou seja,
A estatística T me diz se por exemplo os valores de
Agora, armazenando os mapas em arquivos no formato Analyze (um IMG e um HDR):
f.write.analyze(mapaTcongruente, "./data/MapaCongruente",
pixdim = c(4, 4, 4),
originator = c(23.5, 32.5, 19, 1, 1))
f.write.analyze(mapaTincongruente, "./data/MapaIncongruente",
pixdim = c(4, 4, 4),
originator = c(23.5, 32.5, 19, 1, 1))Um artigo muito interessante que resume o uso de GLM nas análises de fMRI: https://doi.org/10.3389/fnhum.2011.00028