fMRI
A imagem por ressonância magnética funcional (fMRI, do inglês Functional Magnetic Ressonance Imaging), é uma técnica específica do uso da imagem por ressonância magnética (MRI, do inglês Magnetic Resonance Imaging) capaz de detectar variações no fluxo sanguíneo em resposta à atividade neural. A fMRI tem dominado o cenário do mapeamento cerebral em parte devido ao fato de não utilizar radiação ionizante ou contraste exógeno. Além disso, essa técnica possui boa resolução espacial, embora a resolução temporal seja baixa em relação a outras técnicas, como EEG e NIRS.
As mudanças cardiovasculares cerebrais estão relacionadas à atividade elétrica neuronal, devido ao aumento do consumo de oxigênio. O Oxigênio é carregado pela hemoglobina. A hemoglobina desoxigenada (deoxi-Hb) é paramagnética ao passo que a hemoglobina oxigenada (Hb) é diamagnética. Desse modo, a mudança na proporção de Hb/deoxi-Hb durante o aumento de fluxo sanguíneo oxigenado leva a um pequeno aumento do sinal de ressonância subjacente ao local onde ouve aumento da atividade de neurônios. Esse mecanismo é a base do contraste utilizado na maioria dos experimentos de fMRI, e é conhecido como contraste BOLD (do inglês, Blood Oxygenation Level Dependent).
Já em 1936, os químicos Linus Pauling e Charles Coryell mostraram que a hemoglobina possui diferentes momentos magnéticos, dependendo de como seus grupos Hemes estão ligados ao O2. Já em 1990, Seiji Ogawa reconheceu que essa diferença poderia ser usada em MRI para servir de contraste.
EM 1989: Hipotese de Roy & Sherrington: Aumento da atividade metabólica local, por questões de manutenção da homeostase, é seguido de um aumento de fluxo sanguíneo para esta região.
Em um estudo pioneiro, em 1990, Ogawa e colegas avaliaram roedores com um equipamento de MRI de alto campo magnético (7.0 T) e investigaram a hipótese de que a manipulação da proporção os níveis de O2 e CO2 no sangue alteraria o contraste em imagens ponderadas T2*. Eles mostraram redução do sinal próximo a vasos ricos em dHb, que era revertido pelo aumento do fluxo sanguíneo oxigenado através da manipulação da quantidade de gás inalado.
O cérebro é incapaz de armazenar glicose, embora esse seja o único substrato que ele consome. Maior fluxo sanguíneo significa maior disponibilidade de glicose e oxigênio, na forma de hemoglobina oxigenada, em parte devido à dilatação local dos vasos sanguíneos. O aumento do fluxo sanguíneo também acompanha o aumento da atividade elétrica de neurônios. Comumente o aumento do fluxo de oxigênio é maior que o oxigênio consumido na queima de glicose, o que causa a diminuição de dHb nesses vasos sanguíneos. Essas mudanças nas propriedades magnéticas do sangue, fazem com que as áreas ativas interfiram menos na magnetização transversa e no eventual decaimento de T2* induzido por deformação magnética espacial, levando a um leve aumento do sinal de MRI
O parâmetro básico para a resolução temporal é o Tempo de repetição ou TR, que representa o quão frequentemente uma determinada fatia do cérebro é excitada por um pulso RF e decaí ao ponto inicial,, o que no sinal será observado por sua perda de magnetização relativa a T2. TRs podem variar de muito curtos (500 ms) até muito longos (3 s). Especificamente para o fMRI, a resposta hemodinâmica dura em torno de 10 segundos, aumentando multiplicadamente (sempre proporcional ao valor original), atingindo o ápice de 4 a 6 segundos, e então caindo da mesma maneira. Mudanças no fluxo sanguíneo ocorrem via mudanças no sistema vascular concomitantes a atividade neuronal ao longo do tempo. Por essas resposta serem contínuas e suaves, fazer uma amostragem com TRs mais rápidos não iria ajudar. Apenas aumentariam o número de pontos na curva obtidos por uma simples interpolação linear mais não adicionaria informação relevantes aos dados. Paradigmas experimentais tais como um escalonamento quando ocorre a apresentação do estímulo em várias séries de testes podem ajudar a melhorar a resolução temporal, mas reduz o número efetivo de dados pontuais obtidos.
A mudança no sinal MR causado pela atividade neuronal é chamada de resposta hemodinâmica (HDR do inglês Hemodynamic response). O HDR possui um atraso em relação a atividade neuronal entre 1 e 2 segundos, visto que o sistema vascular leva esse tempo para responder a necessidade cerebral de glicose. O pico do sinal da MR normalmente ocorre 5 segundos após o estimulo. Se os neurônios continuarem disparando, o que é chamado de estímulo contínuo, o pico se espalha para um platô enquanto os neurônios se mantiverem ativos. Depois que a atividade cessa, o sinal BOLD cai para os valores originais, os níveis basais, ou podem cair ainda mais em um fenômeno chamado undershoot (supra limiar). A resposta de um único voxel ao longo do tempo representa seu curso temporal. O sinal pode vir de maneira indesejada o que é chamado de ruído e normalmente advém do próprio aparelho de ressonância magnética, da atividade cerebral espontânea, e pode ser tão forte quanto o sinal em si. Para eliminar o ruído estudos que fazem uso do fMRI repetem o estimulo múltiplas vezes para calcular uma média.
Brain Extraction Tool
O algoritmo BET elimina o tecido não cerebral a partir de uma imagem de toda a cabeça. Ele também pode estimar as superfícies interna e externa do crânio e a superfície externa do couro cabeludo, no caso de imagens de entrada T1 e T2 de boa qualidade.
Processamento dos dados e tratamento
Para o tratamento e processamento das imagens, pode-se separar nos seguintes passos:
Suavização espacial (spatial smoothing): borrar a imagem. Muitas vezes, quando trabalhamos com imagens distintas, temos cérebros com formatos distintos. Para padronizar esses dados, utilizamos métodos de deformação de imagem para adição a um modelo padrão. Um modelo comum é o MNI152, deformando para um template, o que chamamos de normalização espacial. Como essa normalização nunca é perfeita, ainda sobram alguns voxels que não ficam perfeitamente alinhados.
normalização espacial
segmentação: separa entre o que for substância branca, substância cinzenta e liquor.
Voxel based morphometry VBM: Baseado nas três etapas e com análise de inferência bayesiana: Para cada voxel, estima a prob (%) de ser substância branca, cinzenta, liquor, etc…
No geral, analisamos mais substância cinzenta no VBM.
John Ashburner indicou que não querem verificar a probabilidade mas sim a redução modulando a deformação e tendo uma estimativa de volume. A VBM é portanto uma medida de diferenças estruturais ou anatómicas entre um grupo de controle clínico e saudável. O VBM permite a análise da concentração de tecido numa base de voxel por voxel. A este respeito, a VBM difere de outras medidas de morfometria que use uma abordagem de região de interesse (ROI). Os dados de VBM são normalizados e mapeados para um modelo cerebral que reduz o impacto das diferenças individuais na anatomia do cérebro. Medidas VBM mostraram diferenças na concentração de substância cinzenta nos lobos temporal e frontal em pacientes com esquizofrenia e controles saudáveis.
Na sua forma mais simples, a morfometria baseada em voxel (VBM) envolve uma comparação voxel-wise (não sei como traduzir) da concentração local de substância cinzenta entre dois grupos de indivíduos. O procedimento envolve a normalização espacial de imagens de alta resolução de todos os participantes do estudo no mesmo espaço estereotáxico. Segue-se a segmentação da massa cinzenta a partir das imagens espacialmente normalizadas e suavização dos segmentos de matéria cinzenta. Testes estatísticos paramétricos de voxel que comparam as imagens de substância cinzenta suavizadas dos dois grupos são realizados. Em seguida, são aplicadas as correções para comparações múltiplas feitas usando a teoria dos campos aleatórios de Gauss. Ashburner, descreve os passos envolvidos no VBM, enfatizando a segmentação da massa cinzenta a partir de imagens RM com artefato não uniformizado e fornecendo avaliações das premissas que sustentam o método, incluindo a precisão da segmentação e as suposições feitas sobre a distribuição estatística dos dados.
A morfometria baseada em deformação e baseada em tensores em referência a métodos para estudar formas cerebrais baseadas em campos de deformação obtidos por registro não-linear de imagens cerebrais. Quando comparamos grupos, a morfometria baseada em deformação (DBM) usa campos de deformação para identificar diferenças nas posições relativas de estruturas dentro dos cérebros dos sujeitos, enquanto usamos o termo morfometria baseada em tensores para nos referirmos àqueles métodos que localizam diferenças na forma local de estruturas cerebrais.
Explicação do Jacobiano do modelo da imagem: Efeito da modulação de imagens segmentadas. O determinante Jacobiano representa as mudanças de volume devido à normalização espacial não linear. Essas alterações de volume são usadas para modular o resultado da segmentação.
O VBM segmenta a imagem entre massa cinzenta, branca e fluído espinal, adequando as imagens para um template e suavizando-as. A análise estatística é baseada no General Linear Model (GLM). O modelo básico constitui na normalização, segmentaçãoe suavização.
Ex: GM-modulated (massa cinzenta modulada)…é o VBM com a modulação pelo jacobiano
Com grupos de indivíduos, tem-se grupos de voxel em vários grupos para cada voxel para identificar a intensidade do GM-modulated em múltiplos indivíduos (normalmente fazem um teste T). Muitos estudos se apoiam no VBM, mas conforme avançam as pesquisas de computação, a modelagem do córtex vem como uma superfície cortical ao invés da massa cinzenta.
No entanto, essa aplicação pode ser demorada e só pode fornecer medidas de áreas bastante amplas. Diferenças menores no volume podem ser negligenciadas uma vez que o VBM registra cada cérebro para um modelo, o que implica em se livrar da maioria das grandes diferenças na anatomia do cérebro entre as pessoas. Então as imagens cerebrais são suavizadas para que cada voxel represente a média de si mesmo e seus vizinhos. Finalmente, o volume da imagem é comparado entre os cérebros em cada voxel.
É importante o cuidado na suavização pois o VBM pode ser sensível a vários artefatos, que incluem desalinhamento das estruturas cerebrais, classificação errônea dos tipos de tecido, diferenças nos padrões de dobra e na espessura cortical. Todos estes erros de classificação podem confundir a análise estatística e diminuir a sensibilidade de verdadeiros efeitos volumétricos ou aumentar a chance de falsos positivos. Para o córtex cerebral, foi demonstrado que as diferenças de volume identificadas com a VBM podem refletir principalmente diferenças na área de superfície do córtex, do que na espessura cortical.
No SPM:
Entre os desenvolvimentos que despertam interesse estão os métodos de análise automática de imagens, que permitem a quantificação de anormalidades em exames radiológicos no nível do voxel (a unidade tridimensional básica das imagens cerebrais, de volumes na ordem de poucos milímetros cúbicos). O método de análise voxel-a-voxel mais conhecido é o Statistical Parametric Mapping (SPM). O plugin SPM para o MATLAB executa diversas etapas no processamento de imagens de RM. Deformação não linear: cada região da imagem ele faz um mapeamento para o template.
No SPM aplica-se algoritmo para smooth a imagem, a convolução basicamente faz uma média ponderada dos vizinhos de cada conjunto de voxels.
Bases físicas, neurológicas e pré-processamento
O tecido pode ser caracterizado por dois tempos diferentes de relaxamento - T1 e T2. No T1 (tempo de relaxamento longitudinal) é a constante de tempo que determina a taxa na qual os prótons excitados retornam ao equilíbrio. É uma medida do tempo gasto para girar prótons para realinharem-se com o campo magnético externo. T2 (tempo de relaxamento transverso) é a constante de tempo que determina a taxa na qual os prótons excitados atingem o equilíbrio ou saem de fase um com o outro. É uma medida do tempo necessário para os prótons girarem e perderem a coerência de fase entre os núcleos girando perpendicularmente ao campo principal.
Para ver estruturas cerebrais, utilizamos: * T1 * T2 * Flair * DWI
O EPI T2*: uma variação para aquisição de dados fMRI. Configuração de dados de forma extremamente sensível.
Obs: DTI para ver tratos/tractografia ou outras análises
Resolução fMRI e comparação:
No caso de fMRI, a coleta está em torno de 1-3s, em geral coleta-se a cada 2s. A tava de amostragem então é a cada 0,5hz, o que é muito mais lento que outras técnicas como EEG, NIRS etc. A resolução temporal é muito inferior ao EEG mas a resolução espacial é melhor que o EEG.
Em ressonância, denominamos por TR o tempo de repetição (tempo entre as imagens da coleta).
Os métodos mais acessíveis, como a tomografia computadorizada (TC), a ressonância magnética (RM) e a tomografia computadorizada por emissão de fóton único (single photon emission computed tomography - SPECT) são recursos importantes para o diagnóstico diferencial de demências; para o diagnóstico e manejo de doenças neurológicas que comumente cursam com sintomas psiquiátricos. No caso, SPECT e PET possuem maior resolução temporal e espacial que fMRI:
Embora a ressonância magnética funcional (fMRI) tenha algumas vantagens sobre PET e SPECT, devido às suas características de melhor resolução espacial e, principalmente, temporal, não-invasiva (sem uso de radiação ionizante), que permite múltiplas seqüências/estudos do mesmo indivíduo na mesma sessão, os modelos para quantificação de informação e a capacidade de estudar uma gama muito maior de funções cerebrais, que incluem principalmente a neurotransmissão e os neurorreceptores, são específicos da tomografia de emissão (PET e SPECT)
Processo de acoplamento hemodinâmico (medida indireta):
- Aumento da atividade neuronal local
- Dilatação das arteríolas
- Aumento do fluxo sanguíneo
- Diminuição da concentração de desoxihemoglobina
- Aumento da sinal BOLD por conseguinte
Pré-processamento das imagens:
Fases do pré-processamento:
Slice-timing correction: Faz interpolação para virtualmente alinhar todas as fatias para o mesmo ponto no tempo para ficar na mesma referência. Maior parte dos programas utiliza método da translação de fase (coleta ascendente, descendente, intercalado)
Correção de movimento/Motion Correction: Ou também images realignment. Translação de corpo rígido. Temos três parâmetros de translação (base X, Y, Z), e três parâmetros de rotação. Por isso chamamos de translação de 6 parâmetros.
Filtros/detrend: Para cada voxel, é aplicado um filtro temporal ao sinal correspondente. Esse filtro em geral pode ser separado em dois tipos (pode variar conforme taxa de amostragem). Se o interesse é obter mapas de ativação, em geral se utiliza um filtro passa-alta. O sinal da ressonância também é sensível a temperatura (não só desoxi). Conforme a máquina funciona e esquenta, queremos eliminar os artefatos de baixa-frequência.
Para análise de conectividade funcional, utiliza-se passa-banda
Outro filtro comum é o detrend: O sinal bold pode estar caindo com o tempo e para isso é aplicado o filtro: aplicando regressão linear menos o predito (linear), é possível ajustar também fazer um quadrático (parábola), já também foi aplicado cúbico na tentativa de limpar os ruídos. Geralmente também pode ser de baixa frequência.
Spatial normalization: Colocar no template específico para todos os sujeitos. Trabalhar com grupos de indivíduos.
Spatial smoothing: Corrigir alguns voxels que não foram bem encaixados e manter as propriedades estatísticas. “Borrar”
Leitura dos dados:
#Carrega a biblioteca
require(AnalyzeFMRI)## Carregando pacotes exigidos: AnalyzeFMRI## Carregando pacotes exigidos: R.matlab## R.matlab v3.6.2 (2018-09-26) successfully loaded. See ?R.matlab for help.##
## Attaching package: 'R.matlab'## The following objects are masked from 'package:base':
##
## getOption, isOpen## Carregando pacotes exigidos: fastICA## Carregando pacotes exigidos: tcltk## Warning in fun(libname, pkgname): couldn't connect to display ":0"#Leitura dos dados
volumes=f.read.volume("./data/Stroop.nii")
#Checar as dimensoes da imagem
dim(volumes)## [1] 45 54 45 180#Plotar uma imagem axial do primeiro volume
image(volumes[,,25,1],col=gray((0:100)/100))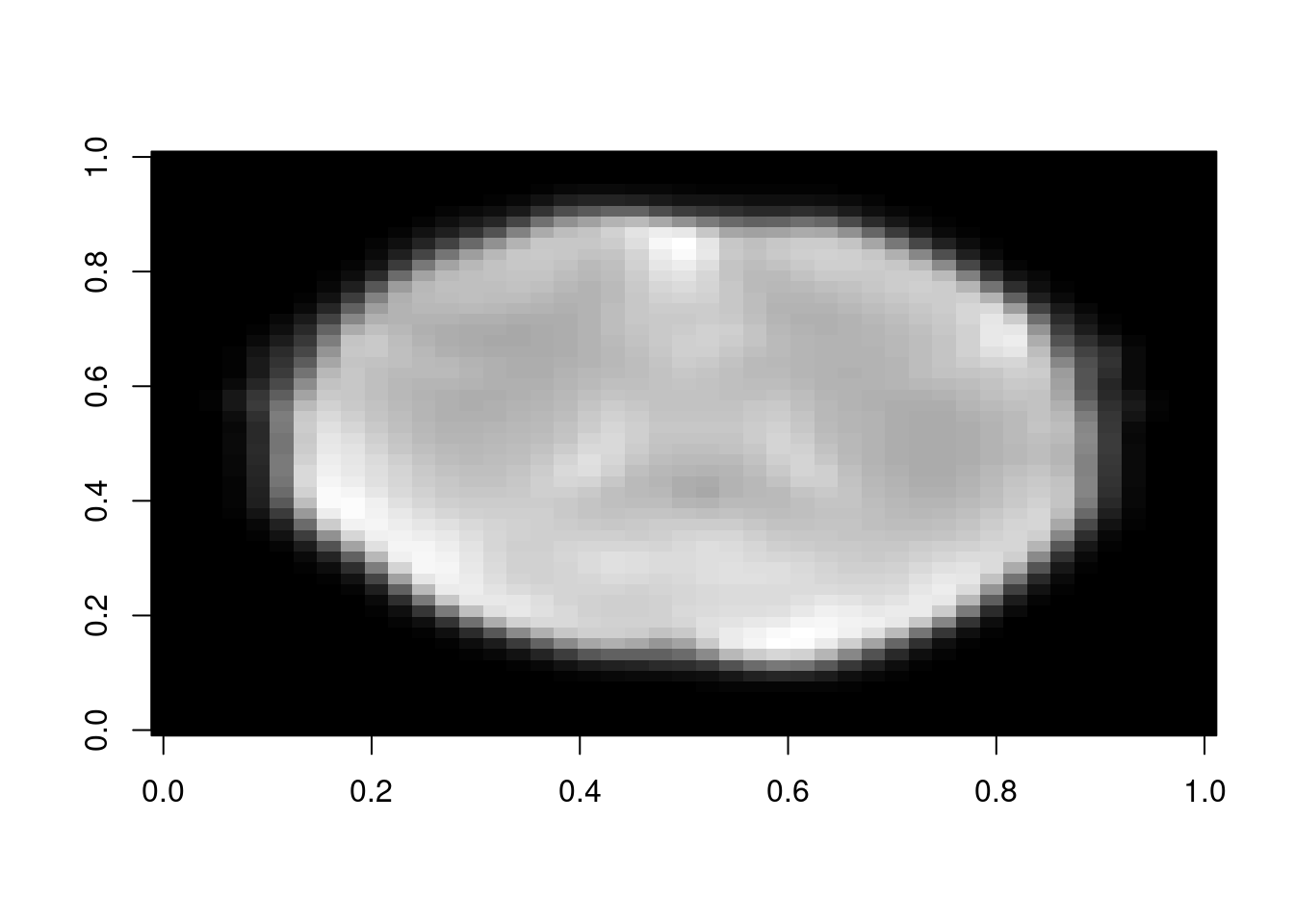
Verificar em todos os pontos do tempo:
#Plotar o sinal BOLD da imagem preprocessada
#no voxel=x=20,y=30,z=25
ts.plot(volumes[20,30,25,])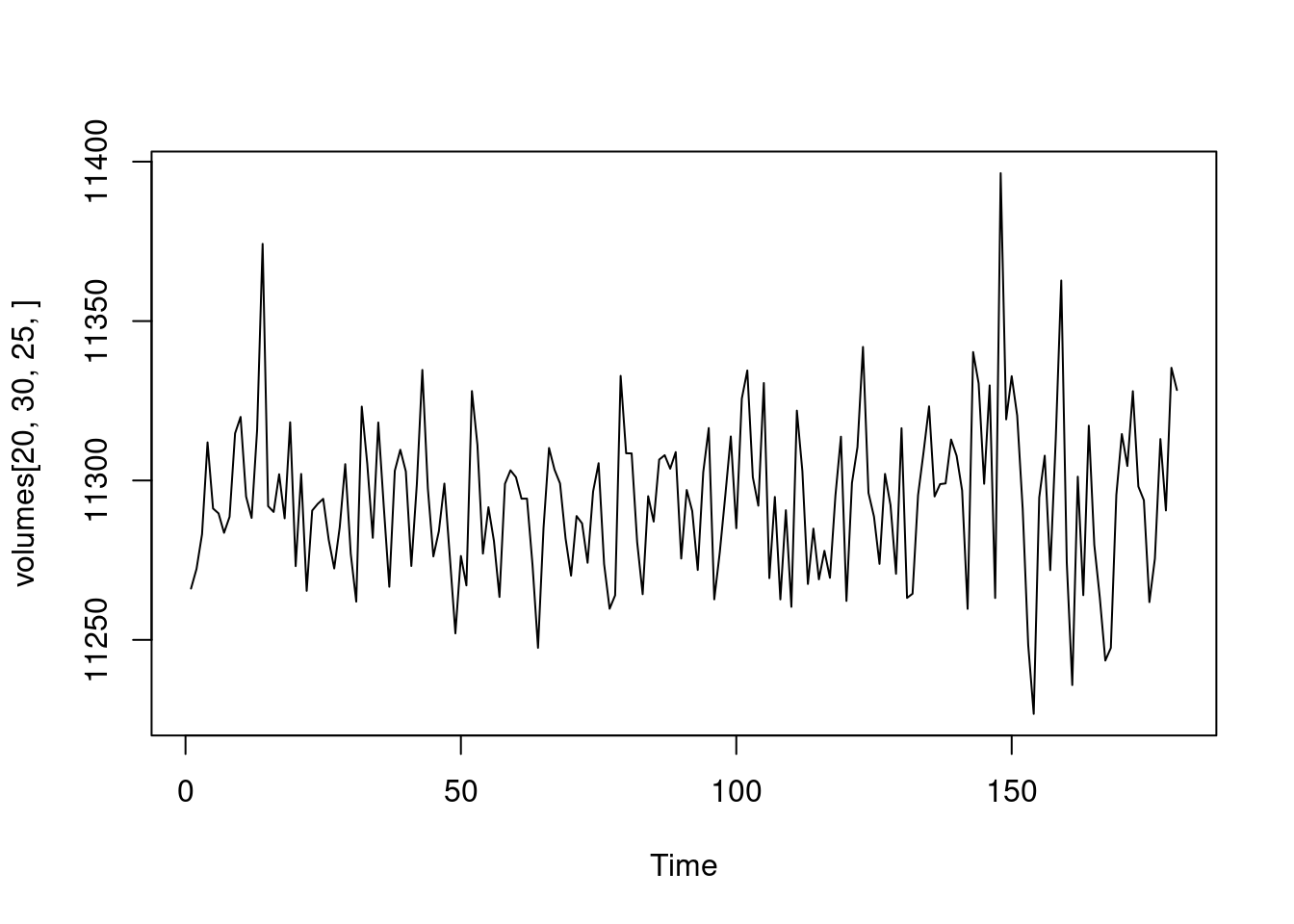
Para marcar a posição do voxel:
volumes[20,30,25,]=0
image(volumes[,,25,1], col=gray((0:100)/100))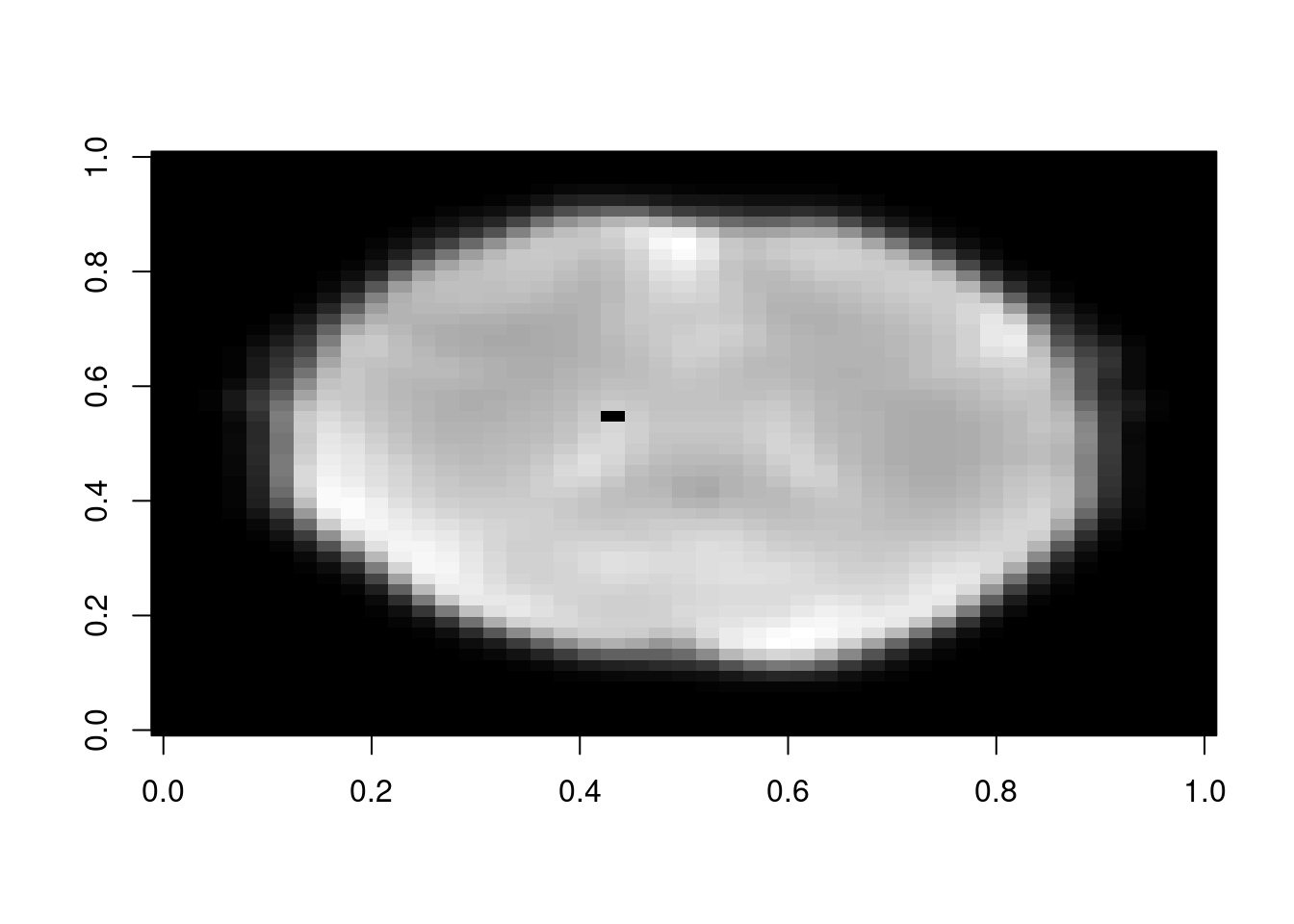
dim(volumes)## [1] 45 54 45 180